2016年04月07日掲載 【コメツキムシのDNAバーコーディング/種は遺伝子だけで判定できるのか?】
DNAバーコーディングとは、遺伝子を使って種同定をするための世界的プロジェクトです。わたしたちは、日本のコメツキムシ科についてバーコードデータベースを作りました。コメツキムシの種同定はとても難しいので、バーコードが今後役立つだろうと思ったからです。ところがそのデータを分析してみると、思いがけず「種は遺伝子だけで判別できるのか」という大きな問題に関わりそうな結果が得られてきました。
なぜコメツキムシなのか
コメツキムシ科は、どの種も特徴に乏しい形と色(だいたい茶色か黒色のモノトーン)をしているので、種同定がとても難しいグループと見なされています。そのくせ、叩き網などではたくさん採れてくるし、農作物に被害をもたらす種も多いので、環境調査や害虫防除に関わる方々はずいぶん手を焼いているようです。ちなみに、私の本当の専門は発光生物です。ところが、中南米にいる発光性コメツキムシの研究に手を出したことがきっかけでコメツキムシの世界に深入りし、とうとう日本のコメツキムシのDNAバーコーディングを手掛けることになってしまいました(日本には発光するコメツキムシが1種もいないというのに!)。今ではすっかりコメツキムシにハマってしまいましたが、種を見分けるのは相変わらず下手くそです。
バーコーディングデータベースを作った
私たちがやったことはシンプルです。日本中からひたすらコメツキムシを集めて、バーコードプロジェクトで定められた遺伝子領域をシーケンスします。もちろん、解析に使った個体はコメツキムシ分類の専門家にすべて種同定してもらいました。集めたデータは、標本の写真とともに国際DNAバーコーディングデータベース(BOLD)に登録します。これで完成。日本で普通に見つけることのできる種はほぼ網羅していますし、BOLDを通じて世界中から誰でも自由にこのデータベースにアクセスできますので、日本のコメツキムシの種同定を必要とする人たちの役に立つはずです。なにより、コメツキムシに興味を持ってしまったけれど種判別が苦手な私自身にとって大いに役立ちそうです。
バーコーディングはすごい
DNAバーコーディングのオリジナルコンセプトは、遺伝子情報だけで世界中の生物を種同定できるようにするという野心的なものでした。ところが、生物の種とはそんな単純に決められるものではないという反論も多く、このコンセプトは次第に弱められ「バーコーディングは、種を同定するための補助ツールになる」というくらいのところに意見はまとまりかけているのが現状です。それでも、DNAバーコーディングがとても役に立つツールであることには変わりがありません。あまり詳しくない分類群の種同定をしようとして検索図鑑を調べてみて、いきなり最初の方の検索キーでつまずいた経験を持つ人も多いはず。それがデータベースさえしっかりしていれば、ちょっと遺伝子解析するだけで「あの種かこの種」という最後の検索キーあたりまで辿り着くのですから、すごいことです。
遺伝子だけで種が分かるのか?
ところが、DNAバーコーディングの生みの親であるゲルフ大学のエベールらは、もともとの「遺伝子だけで種がわかるはず」という野望を捨てていなかったようです。じつは、これまでにもそうした巻き返しの動きはいくつかありましたが、最近とうとうBOLDに「遺伝子だけで種を判別するツール」BIN(Barcoding Index Number)システムが実装されたのです。BINシステムは、遺伝子のホモロジーが何%あったら同種だとか別種だとか単純に決定するものではなく、既存の蓄積データから経験的反復サーチによって種を判断する高度なシステムです。これを使えば、約90%の種が正しく判定できるということが、昆虫、魚、鳥などで確かめられていました。そこで、わたしたちも自分たちが解析したコメツキムシのデータを使ってBIN解析を行ってみました。
種とBINが一致しない
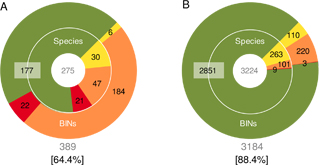
図3: 円グラフAは日本産コメツキムシ科バーコードデータ(275種/389 BINs)、円グラフBは欧米を中心とした昆虫、魚類、鳥などの8つのバーコードデータ(3224種/3184 BINs)を合計したもの(Ratnasingham & Hebert, 2013より)のBIN解析結果
緑色はMATCHES(種とBINが一対一対応)、黄色はMERGES(複数種が1BINにまとまる)、橙色はSPLITS(1種が複数のBINに分かれる)、赤色はMIXTURE(MERGESとSPLITSの混合)。例えばAの橙色は、コメツキムシの47種が184 BINsに分けられたことを表している。Bでは、全体の88.4%(2851/3224)の種がBINと一対一対応しているが、Aでは全体の64.4%(177/275)の種しかBINと一対一対応していない。詳しくはOba et al. 2015を参照。
(クリックで拡大します)
その結果は、まったく意外なものでした。実際の種とBINシステムが分けた種が、全体の64%しか一致しなかったのです。つまり残りの4割は、ほんとうは1種なのにBINが複数種であると判断したか、ほんとうは複数種なのにBINが1種と判断したか、もしくはその混合でした。なぜこんなことになったのでしょう?
「日本のコメツキムシの分類が未熟だっただけじゃないか」という意見もあるでしょう。しかし、私にはそれほど単純な理由だとは思えません。確かに、日本のコメツキムシ分類には、まだ議論の余地のある種が存在するのも事実ですが、どれも世界基準の分類システムに厳密に準じて時間をかけて決定されてきた種なのですから。
むしろ個人的には、どちらかというと日本のコメツキムシは種を細かく分け過ぎかなと思っていたのですが、種とBINが一致しなかったものの多くが「一種のはずがBINでは複数種」と判断されたものだったことは意外でした。つまり、BINの基準では、かなりの種が「もっと細かく分けるべきだ」と判定されたのです。
遺伝子だけで種が分かるのか?
BINと種の間に生じたこの矛盾について、現在わたしは、欧米と日本の生物多様性の高さの違いがその原因ではないかと考えています。たとえば、日本には600種以上のコメツキムシが知られていますが、似たような国土面積を持つイギリスにはたった73種類しかいません。また、広大な北米全体を合わせても、日本と同程度の種数です。このような多様性が大きく違う生物相を同じ分類基準で扱ってきたことに、そもそもの問題があったのではないでしょうか。
実際、DNAバーコーディングは、これまで生物多様性の比較的乏しい欧米の生物を中心にデータ集積が行われてきました。BINのアルゴリズムも、とうぜん基本的にこれら欧米種のデータに基づいて構築されています。一方、日本を含む生物多様性の高い地域(多様性ホットスポットと呼ばれる地域)でのバーコード収集は十分に進んでいるとは言えません。
結論を述べると、少なくともある分類群においては「生物多様性の高い地域の生物相では、多様性の低い欧米とは種と種の区切り基準が異なっているのではないか」というのが私の仮説です。このあたりの大胆な議論は、論文の中には十分に盛り込めていませんが、日本でも今後ひとつの分類群をある程度網羅するような大規模なDNAバーコーディングがもっと行われれば、他の分類群でもBINとの矛盾がたくさん見つかってくるのではないかと想像しています。そうだとすると、「遺伝子だけで種が分かる」という野望はもう少し慎重に考えなければならないということになりそうです。
おわりに
「日本の生物は分類が難しい」という話を耳にすることがありますが、もしかすると、それにはここで紹介したような「欧米の分類基準が日本では通用しない」という根本的な理由があるのかもしれません。一見地味に見えるDNAバーコーディングデータベース作りから、ホットスポットの生物多様性という大きなテーマが見えてくることを夢見ています。
参考文献
- Oba Y, Ôhira H, Murase Y, Moriyama A, Kumazawa Y (2015) DNA barcoding of Japanese click beetles (Coleoptera, Elateridae). PLOS ONE 10: e0116612. doi: 10.1371/journal.pone.0124857
- Ratnasingham S, Hebert PDN (2013) A DNA-based registry for all animal species: the Barcode Index Number (BIN) system. PLOS ONE 8: e66213. doi:10.1371/journal.pone.0066213
« カイコで遺伝子を強力に働かせることに成功 | トピック一覧 | トビイロウンカに強いイネが持つ抵抗性遺伝子の正体 »
応用動物学/昆虫学最新トピック
プロの研究者でもまだ知らないような、出来たてホヤホヤの最新研究成果を分かりやすくお伝えします。
日本応用動物昆虫学会(応動昆)
「むしむしコラム・おーどーこん」は、日本応用動物昆虫学会電子広報委員会が管理・運営しています。

